如果有一個包含數十個甚至數百個特征的數據集,每個特征都可能對你的機器學習模型的性能有所貢獻。但是并不是所有的特征都是一樣的。有些可能是冗余的或不相關的,這會增加建模的復雜性并可能導致過擬合。特征重要性分析可以識別并關注最具信息量的特征,從而帶來以下幾個優勢:
- 改進的模型性能
- 減少過度擬合
- 更快的訓練和推理
- 增強的可解釋性
下面我們深入了解在Python中的一些特性重要性分析的方法。
特征重要性分析方法
1、排列重要性 PermutationImportance
該方法會隨機排列每個特征的值,然后監控模型性能下降的程度。如果獲得了更大的下降意味著特征更重要。
from sklearn.datasets import load_breast_cancer
from sklearn.ensemble import RandomForestClassifier
from sklearn.inspection import permutation_importance
from sklearn.model_selection import train_test_split
import matplotlib.pyplot as plt
cancer = load_breast_cancer()
X_train, X_test, y_train, y_test = train_test_split(cancer.data, cancer.target, random_state=1)
rf = RandomForestClassifier(n_estimators=100, random_state=1)
rf.fit(X_train, y_train)
baseline = rf.score(X_test, y_test)
result = permutation_importance(rf, X_test, y_test, n_repeats=10, random_state=1, scoring='accuracy')
importances = result.importances_mean
# Visualize permutation importances
plt.bar(range(len(importances)), importances)
plt.xlabel('Feature Index')
plt.ylabel('Permutation Importance')
plt.show()
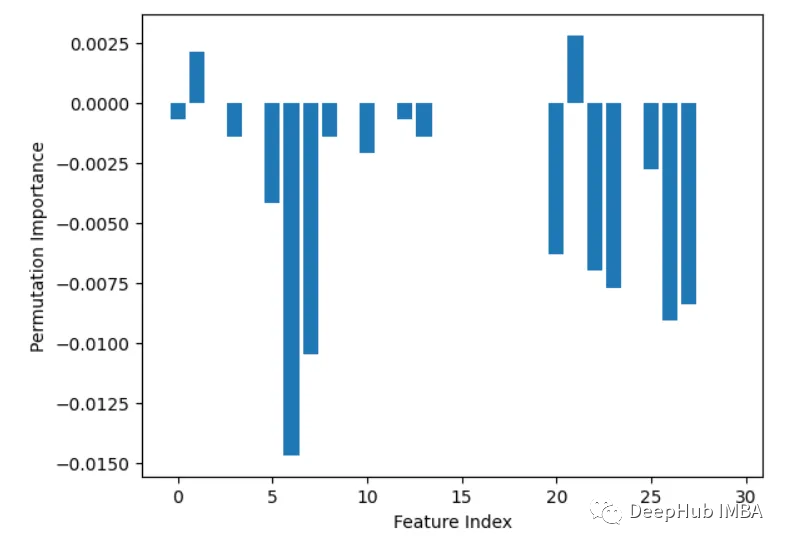
2、內置特征重要性(coef_或feature_importances_)
一些模型,如線性回歸和隨機森林,可以直接輸出特征重要性分數。這些顯示了每個特征對最終預測的貢獻。
from sklearn.datasets import load_breast_cancer
from sklearn.ensemble import RandomForestClassifier
X, y = load_breast_cancer(return_X_y=True)
rf = RandomForestClassifier(n_estimators=100, random_state=1)
rf.fit(X, y)
importances = rf.feature_importances_
# Plot importances
plt.bar(range(X.shape[1]), importances)
plt.xlabel('Feature Index')
plt.ylabel('Feature Importance')
plt.show()
3、Leave-one-out
迭代地每次刪除一個特征并評估準確性。
from sklearn.datasets import load_breast_cancer
from sklearn.model_selection import train_test_split
from sklearn.ensemble import RandomForestClassifier
from sklearn.metrics import accuracy_score
import matplotlib.pyplot as plt
import numpy as np
# Load sample data
X, y = load_breast_cancer(return_X_y=True)
# Split data into train and test sets
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.3, random_state=1)
# Train a random forest model
rf = RandomForestClassifier(n_estimators=100, random_state=1)
rf.fit(X_train, y_train)
# Get baseline accuracy on test data
base_acc = accuracy_score(y_test, rf.predict(X_test))
# Initialize empty list to store importances
importances = []
# Iterate over all columns and remove one at a time
for i in range(X_train.shape[1]):
X_temp = np.delete(X_train, i, axis=1)
rf.fit(X_temp, y_train)
acc = accuracy_score(y_test, rf.predict(np.delete(X_test, i, axis=1)))
importances.append(base_acc - acc)
# Plot importance scores
plt.bar(range(len(importances)), importances)
plt.show()
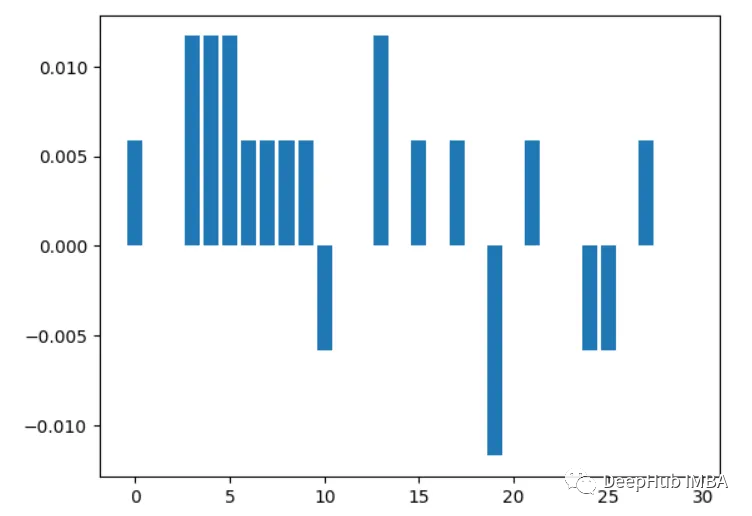
4、相關性分析
計算各特征與目標變量之間的相關性。相關性越高的特征越重要。
import pandas as pd
from sklearn.datasets import load_breast_cancer
X, y = load_breast_cancer(return_X_y=True)
df = pd.DataFrame(X, columns=range(30))
df['y'] = y
correlations = df.corrwith(df.y).abs()
correlations.sort_values(ascending=False, inplace=True)
correlations.plot.bar()

5、遞歸特征消除 Recursive Feature Elimination
遞歸地刪除特征并查看它如何影響模型性能。刪除時會導致更大下降的特征更重要。
from sklearn.ensemble import RandomForestClassifier
from sklearn.feature_selection import RFE
import pandas as pd
from sklearn.datasets import load_breast_cancer
import matplotlib.pyplot as plt
X, y = load_breast_cancer(return_X_y=True)
df = pd.DataFrame(X, columns=range(30))
df['y'] = y
rf = RandomForestClassifier()
rfe = RFE(rf, n_features_to_select=10)
rfe.fit(X, y)
print(rfe.ranking_)
輸出為[6 4 11 12 7 11 18 21 8 16 10 3 15 14 19 17 20 13 11 11 12 9 11 5 11]
6、XGBoost特性重要性
計算一個特性用于跨所有樹拆分數據的次數。更多的分裂意味著更重要。
import xgboost as xgb
import pandas as pd
from sklearn.datasets import load_breast_cancer
import matplotlib.pyplot as plt
X, y = load_breast_cancer(return_X_y=True)
df = pd.DataFrame(X, columns=range(30))
df['y'] = y
model = xgb.XGBClassifier()
model.fit(X, y)
importances = model.feature_importances_
importances = pd.Series(importances, index=range(X.shape[1]))
importances.plot.bar()

7、主成分分析 PCA
對特征進行主成分分析,并查看每個主成分的解釋方差比。在前幾個組件上具有較高負載的特性更為重要。
from sklearn.decomposition import PCA
import pandas as pd
from sklearn.datasets import load_breast_cancer
import matplotlib.pyplot as plt
X, y = load_breast_cancer(return_X_y=True)
df = pd.DataFrame(X, columns=range(30))
df['y'] = y
pca = PCA()
pca.fit(X)
plt.bar(range(pca.n_components_), pca.explained_variance_ratio_)
plt.xlabel('PCA components')
plt.ylabel('Explained Variance')

8、方差分析 ANOVA
使用f_classif()獲得每個特征的方差分析f值。f值越高,表明特征與目標的相關性越強。
from sklearn.feature_selection import f_classif
import pandas as pd
from sklearn.datasets import load_breast_cancer
import matplotlib.pyplot as plt
X, y = load_breast_cancer(return_X_y=True)
df = pd.DataFrame(X, columns=range(30))
df['y'] = y
fval = f_classif(X, y)
fval = pd.Series(fval[0], index=range(X.shape[1]))
fval.plot.bar()
9、卡方檢驗
使用chi2()獲得每個特征的卡方統計信息。得分越高的特征越有可能獨立于目標。
from sklearn.feature_selection import chi2
import pandas as pd
from sklearn.datasets import load_breast_cancer
import matplotlib.pyplot as plt
X, y = load_breast_cancer(return_X_y=True)
df = pd.DataFrame(X, columns=range(30))
df['y'] = y
chi_scores = chi2(X, y)
chi_scores = pd.Series(chi_scores[0], index=range(X.shape[1]))
chi_scores.plot.bar()
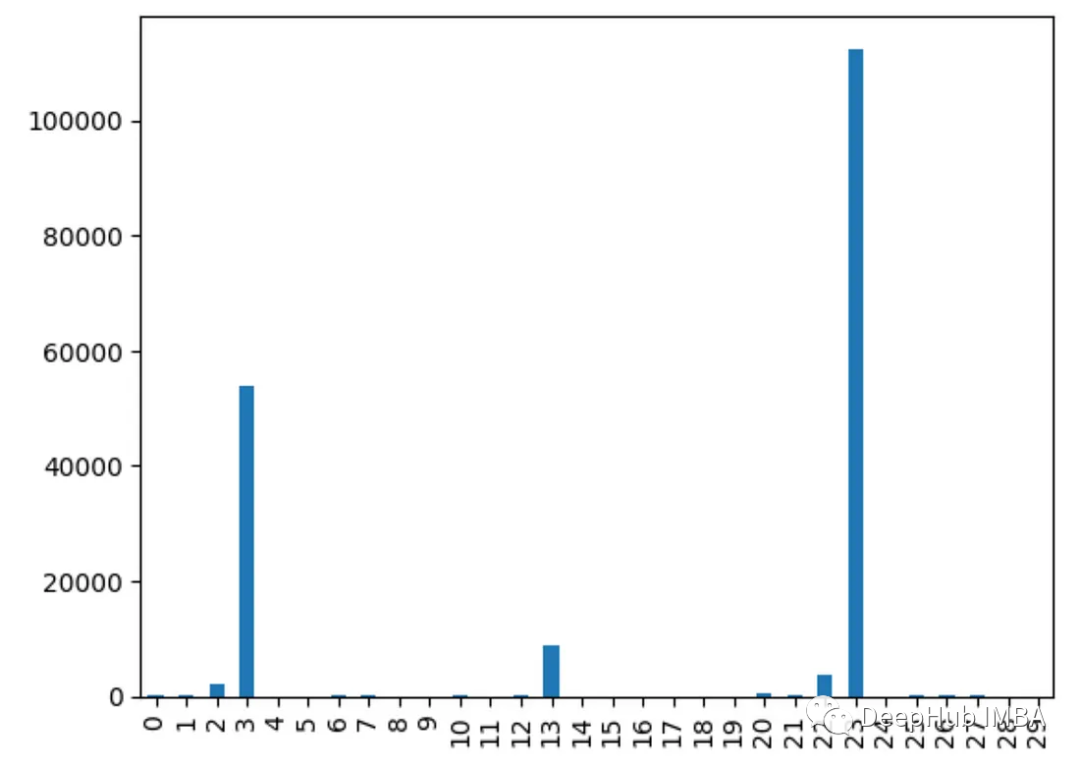
為什么不同的方法會檢測到不同的特征?
不同的特征重要性方法有時可以識別出不同的特征是最重要的,這是因為:
1、他們用不同的方式衡量重要性:
有的使用不同特特征進行預測,監控精度下降
像XGBOOST或者回國模型使用內置重要性來進行特征的重要性排列
而PCA著眼于方差解釋
2、不同模型有不同模型的方法:
線性模型傾向于線性關系、樹模型傾向于接近根的特征
3、交互作用:
有的方法可以獲取特征之間的相互左右,而有一些則不行,這就會導致結果的差異
4、不穩定:
使用不同的數據子集,重要性值可能在同一方法的不同運行中有所不同,這是因為數據差異決定的
5、Hyperparameters:
通過調整超參數,如PCA組件或樹深度,也會影響結果
所以不同的假設、偏差、數據處理和方法的可變性意味著它們并不總是在最重要的特征上保持一致。
-
模型
+關注
關注
1文章
3419瀏覽量
49483 -
機器學習
+關注
關注
66文章
8461瀏覽量
133438 -
python
+關注
關注
56文章
4813瀏覽量
85319 -
數據集
+關注
關注
4文章
1212瀏覽量
25006
發布評論請先 登錄
相關推薦




 Python中進行特征重要性分析的9個常用方法
Python中進行特征重要性分析的9個常用方法











評論