提起達爾文的生物進化論,在人們的普遍認知中,這是開創現代科學的重要理論之一。像地球上其他所有為生存而掙扎的生物一樣,病毒也會進化或變異。讓我們看看人類病毒的來源——蝙蝠病毒的RNA核苷酸序列片段:AAAAT CAAA GCTT GTGTT GAA GAA GTTACAA CAACTCT GGAAG AAACTAAGTT與一小段人類的新型冠狀病毒肺炎(Corona Virus Disease 2019,COVID-19)的RNA核苷酸序列:AAAAT TAAG GCTT GCATT GAT GAG GTTACCA CAACACT GGAAG AAACTAAGTT顯然,冠狀病毒已經改變了它的內部結構以適應新的宿主物種(更準確地說,大約20%的冠狀病毒內部結構都發生了突變),但仍然保持了足夠數量的一致,使它仍然忠于它的起源物種。事實上,研究表明,COVID-19會不斷發生變異,以提高其存活率。在與冠狀病毒的對抗中,我們不僅需要探究擊敗病毒的方法,更需要明白病毒是如何變異的,以及如何應對病毒變異。這篇文章中將從以下幾個方面進行闡述:①從表面上解釋RNA核苷酸序列是什么②使用K-Means創建基因組信息集群③使用PCA實現可視化集群什么是基因組序列?DNA是脫氧核酸的簡稱,其基本單位是脫氧核糖核苷酸(也叫脫氧核苷酸),是大多數生物的遺傳物質,在真核生物、原核生物、DNA病毒內都存在的一種核酸;RNA則是核糖核酸的簡稱,其基本單位是核糖核苷酸,是RNA病毒的遺傳物質。新型冠狀病毒的基因序列就是RNA.基因組測序,通常被比作“解碼”,是分析取自樣本的脫氧核糖核酸(DNA)的過程。在每個正常細胞中有23對染色體,DNA的結構是這樣的:
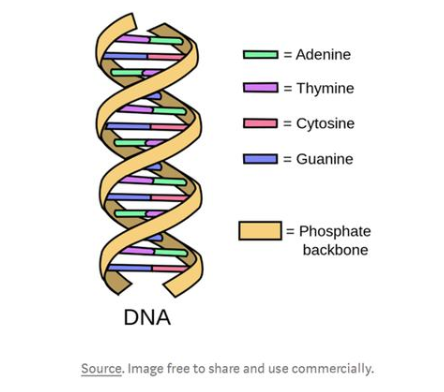
DNA卷曲的雙螺旋結構可以使它展開成階梯狀,這個梯子是由成對的化學字母組成的,叫做堿基。在DNA中有四種堿基:腺嘌呤、胸腺嘧啶、鳥嘌呤和胞嘧啶。腺嘌呤只與胸腺嘧啶結合,鳥嘌呤只與胞嘧啶結合,這些堿基分別用A、T、G和C表示。這些堿基形成了各種各樣的代碼,指導有機體如何構建蛋白質——這就是DNA如何控制病毒一舉一動的基礎。

使用專門的設備,包括測序儀器和專門的標簽,可以顯示特定的DNA序列片段。由此獲得的信息將經過進一步的分析和比較,使研究人員能夠識別基因的變化,與疾病和表型的關系,并確定潛在的藥物靶標。一長串的基因組序列A、T、G和C,代表了有機體對環境的反應,而生物體的突變又是通過改變DNA產生的,因此觀察基因組序列是分析冠狀病毒突變的有效手段,其中序列對齊法是常用的方法,主要通過將兩個或多個核酸序列或者蛋白質序列進行對比,并將其中相似的結構區域突出顯示。序列對齊:給定兩個DNA序列A和B,對齊的方式是將空格分別插入到A和B序列中,得到具有相同長度的對齊后的序列C和D;空格可以插入到任意的位置(包括兩端),但是相同位置不能同時為空格,也即是不存在C[i]和D[i]同時為空格的情況。然后為對齊后的序列的每個位置打分,總分為每個位置得分之和,具體的打分規則如下:a、如果C[i] == D[i]且都不是空格,得3分;b、如果C[i] != D[j]且都不是空格,得1分;c、如果C[i] 或者D[i]是空格,得0分。求給定原序列A和B的一個對齊方案,使得該對齊方案的總分最高。例如,序列原序列A和B如下:String strA = “GATC”; String strB = “ATCG”;則其中一個對齊方案如下:GATC**ATCG該方案總得分score=2*0+3*3 = 9分。因此,經常通過序列對齊方式來比較序列與已知(尤其是功能和結構已知的序列)之間的同源性,預測未知序列的功能。因此本文后續對于序列的分析主要是針對序列對齊后形成的指標特征進行探索和分析。數據的獲取數據可以在Kaggle上找到,如下圖所示:

每一行代表蝙蝠病毒的一個突變。首先,花一分鐘來欣賞大自然是多么不可思議——在幾周內,冠狀病毒已經產生了262個突變來增加存活率。一些重要的列名解釋:
query acc.ver表示原始的病毒標識符。
subject acc.ver是病毒突變的標識符。
% identity表示序列中與原始病毒相同的百分比。
Alignment length表示序列中有多少項是相同的或對齊的。
mismatches表示突變項和原始項之間的不同項數。
bit score代表了一個衡量標準,衡量序列的對齊程度;分數越高,對齊程度越高。
每一列的統計度量如下所示(這些可以在Python中運用data.describe()語句被方便地調用):

有趣的是,通過查看% identity列,我們可以看到一個突變與原始病毒的最小對齊比率約為77.6%。然而巨大的標準偏差(7%的% identity)意味著原始病毒存在廣泛的變異范圍。在bit score中巨大標準偏差證實可以證實這一點——標準偏差大于平均值(即代表變異系統大于1,進一步說明了突變發生情況的多樣性)!通過相關性熱力圖可以很好的呈現變量之間的相關性,圖形中每個單元表示一個特征與另一個特征的相關性。

我們不難發現,很多數據都是高度相關的,這是可以解釋的,因為大多數的度量彼此存在一定的依賴性,因此導致變量之間存在高相關性,可以發現alignment length與bit score之間就具有高度相關性(0.94)。
使用K-Means來創建突變集群K-Means是一種聚類算法,是通過機器學習的方式在特征空間中確定數據點相似群組。我們運用K-Means的目標是找到突變的群體,這樣我們就可以對突變的本質以及如何針對性的處理它們有深入的了解。在此之前,我們首先需要確定集群k的數量,雖然這就像在二維空間中繪制一個點一樣簡單,但在高維空間中是幾乎無法實現的(如果我們想要保留最多的信息)。若用“肘部法則”來選擇k會顯得過于主觀,且不準確,所以我們會用輪廓法來代替。輪廓法是給不同取值k的集群打分,來區分聚類的結果好壞程度(好的聚類:內密外疏,同一個聚類內部的樣本要足夠密集,不同聚類之間樣本要足夠疏遠)。Python中的sklearn庫將使K-Means和輪廓法的實現變得非常簡單。
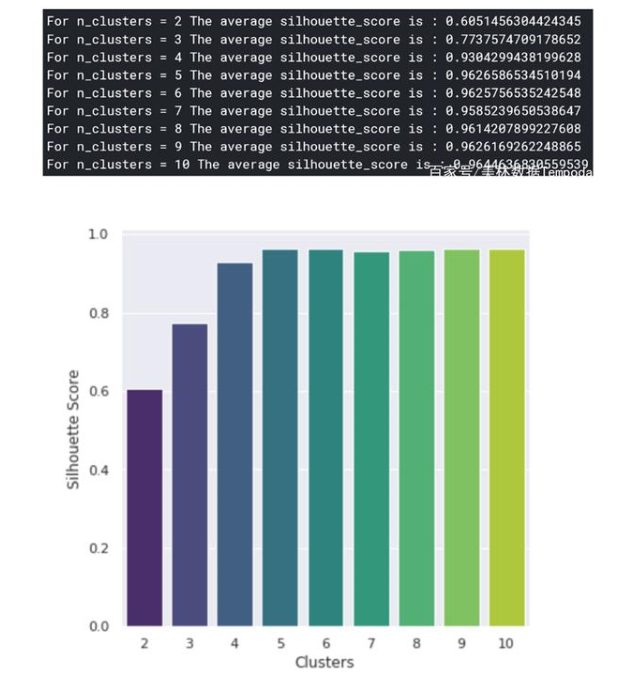
通過對上圖進行分析,可以發現群體數為5時聚類效果最佳。現在,我們可以進一步確定群體中心,這些點是每個群體的中心,代表了不同群體的突變樣本的共性特征。

注:特征已經被標準化,列與列之間無可比性
在此熱力圖中,行:代表不同的群體,列:代表每個群體的屬性。因為在聚類之間需要對于特征按比例進行縮放,以減少不同特征尺度差異的影響,所以圖中的數值在數量(縮放值,非原始尺度下的值)上沒有任何意義,但是,我們可以通過比較每個列中的縮放值,這使得我們可以對每個突變群體的特征相對大小產生一個更直觀的感覺。通過對以上聚類結果的分析,可以讓科學家將更多精力聚焦在對不同突變群體的特征研究上,進而針對性的研究不同類型的疫苗,治療和預防也將變的更有目標性。聚類的結果已經可以幫助我們解決很多方面的問題,但由于存在高維特征及特征之間相關性的存在,讓我們不能更好的去解讀聚類結果,因此,在下一節中,我們將使用PCA來實現聚類結果的可視化呈現。利用PCA進行集群可視化主成分分析是一種降維方法。它選擇多維空間中的正交向量來表示坐標軸,通過特征的空間變換,可以有效降低特征之間的相關性,進而通過貢獻度來保留最多的信息的特征,實現降維目的。同樣,我們可以通過Python的sklearn庫,PCA的執行可以被兩行代碼實現。首先,我們可以檢查被解釋的方差比(explained variance ratio),這是從原始數據集中保留的統計信息的百分比。在本例中,被解釋的方差比是0.9838548580740327,代表信息只有很少部分遺失!在此我們可以確信,無論我們從PCA得到什么分析,數據都是具有很高的可信度。每個新的特征(主成分)都是其他幾個列的線性組合。通過熱力圖,我們可以直觀地了解每一特征對于兩個成份(新的特征)中的重要性。

通過以上圖中數值的分析,關鍵是要理解在成分1中出現高數值是什么意思——在這種情況下,它的特點是有著更高的一致性,即更接近原始病毒;成分2的主要的特點是擁有更低的一致性,即突變遠離原始值,這也反映在bit score的較大差異上。

通過主成分將所有樣本映射到2維空間體系下,可以很明顯發現,病毒突變有5條主線,以下通過對這5條線的分析,可以讓我們獲取更多的信息。可以發現,有四個病毒突變在第一主成分(X軸)的左邊,一個在右邊。第一主成分的特征是alignment length具有很高的取值,這意味著第一個主成分的值越高,對應的alignment length就越長(越接近原始病毒)。因此,第一主成分的低值區與原始病毒的遺傳距離較遠,即大多數病毒集群與原始病毒有很大不同。因此,試圖研制疫苗的科學家應該意識到,這種病毒會發生大量變異。第二主成分(Y軸)在同一群體之間的差異性很小,在不同群體之間明顯分為3個區段,這就需要后續我們進一步分析,以便能夠更好的對于突變群體進行深入了解。結論本文一方面通過使用K-Means聚類算法,能夠幫助我們從眾多突變樣本中快速識別冠狀病毒的五個主要典型突變群體,另一方面用PCA分析方法在二維空間中實現這些群體的可視化展現,通過展示結果可以很直觀的呈現冠狀病毒有很高的突變率(這可能就是它如此致命的原因),通過對于這些分析結果,對于研制冠狀病毒疫苗的科學家來說,可以利用群體的共性特征值結合領域專業知識來充分解讀每個群體的特征信息,以便有針性的、更好的指導疫苗的研制及預防工作。
-
DNA
+關注
關注
0文章
243瀏覽量
31108 -
K-means
+關注
關注
0文章
28瀏覽量
11324
發布評論請先 登錄
相關推薦




 機器學習告訴你,新型冠狀病毒接下來將如何變異?
機器學習告訴你,新型冠狀病毒接下來將如何變異?











評論