領先的制藥公司、生物技術初創企業和前沿生物研究人員正在使用 NVIDIA BioNeMo LLM 服務和框架來開發用于生成、預測和理解生物分子數據的 AI 應用。
科學家們致力于探索 DNA、蛋白質和其他生命體的“基礎構建”,以期獲得全新的洞察,而今日在 NVIDIA GTC 上發布的 NVIDIA BioNeMo 框架將為其研究提供加速。
NVIDIA BioNeMo 框架用于訓練和部署超算規模的大型生物分子語言模型,幫助科學家更好地了解疾病,并為患者找到治療方法。該大型語言模型(LLM)框架將支持化學、蛋白質、DNA 和 RNA 數據格式。
NVIDIA BioNeMo 是 NVIDIA Clara Discovery 藥物研發框架、應用和 AI 模型集的一部分。
正如 AI 通過 LLM 來學習如何理解人類語言,該框架也在學習生物學和化學語言。NVIDIA BioNeMo 使基于生物分子數據的大規模神經網絡訓練更為輕松,助力研究人員發現生物序列中的新模式并獲得新洞察。研究人員可將這些洞察與生物特性或功能乃至人類健康狀況聯系起來。
NVIDIA BioNeMo 框架使科學家能夠使用更大的數據集來訓練大規模語言模型,打造出性能更強大的神經網絡。NVIDIA 的 GPU 優化軟件中心——NVIDIA NGC 上將提供該框架的搶先體驗。
除語言模型框架之外,NVIDIA BioNeMo 還提供一項云 API 服務,該服務將支持越來越多的預訓練 AI 模型。
BioNeMo 框架支持更大的模型
和更準確的預測
如今,使用自然語言處理模型來處理生物數據的科學家一般會訓練相對較小、需要自定義預處理的神經網絡。通過 BioNeMo,科學家可將其擴展為具有數十億參數的 LLM,捕捉分子結構、蛋白質溶解度等信息。
BioNeMo 是 NVIDIA NeMo Megatron 框架的擴展,可實現大規模自監督語言模型的 GPU 加速訓練。這一針對特定領域的框架支持以 SMILES 化學結構標記表征的分子數據、以及以 FASTA 氨基酸和核酸序列字符串表征的分子數據。
OpenFold 聯盟創始成員、哥倫比亞大學系統生物學系助理教授 Mohammed AlQuraishi 表示:“該框架使整個醫療和生命科學行業的研究人員都能利用快速增長的生物和化學數據集。這樣就能更輕松地發現和設計出精準針對疾病分子特征的治療方法。”
BioNeMo 服務提供用于化學和生物學的 LLM
NVIDIA BioNeMo LLM 服務將為希望快速掌握數字化的生物學和化學領域應用的 LLM 的開發者提供四個預訓練語言模型。這些模型針對推理進行了優化,并將通過 NVIDIA DGX Foundry 上運行的云端 API 提供搶先體驗。
ESM-1:這一最初由 Meta AI Labs 發布的蛋白質 LLM 能夠處理氨基酸序列,最終生成用于預測各種蛋白質特性和功能的表征。它還提高了科學家理解蛋白質結構的能力。
OpenFold:這是由學術界和產業界共同成立的 Openfold 聯盟創建的 sota 蛋白質建模工具,它將可以通過 BioNeMo 服務提供其開源 AI 工作流程。
MegaMolBART:這一基于 14 億分子訓練而成的生成式化學模型可用于反應預測、分子優化和新分子的生成。
ProtT5:該模型是在慕尼黑工業大學 RostLab 的帶領下合作開發的,NVIDIA 也是該項目的參與者之一。PortT5 將 ESM-1b 等蛋白質 LLM 的功能擴展到序列生成。
未來,使用 BioNeMo LLM 服務的研究人員將能夠通過 fine-tuning 以及 p-tuning 等新技術,在幾小時內完成 LLM 模型的自定義,提高應用的準確性。相比原來動輒數百萬個樣本的數據集,p-tuning 訓練方法只需要包含幾百個樣本的數據集。
初創企業、研究人員和制藥公司
紛紛采用 NVIDIA BioNeMo
生物技術和制藥行業的專家正在采用 NVIDIA BioNeMo,為新藥研發提供支持。
阿斯利康和 NVIDIA 使用 Cambridge-1 超級計算機開發了 BioNeMo LLM 服務中的 MegaMolBART 模型。這家全球生物制藥公司將使用 BioNeMo 框架,在涵蓋小分子和蛋白質的數據集上訓練一些全球最大的語言模型。該數據集日后還將涵蓋 DNA。
麻省理工學院和哈佛大學旗下博德研究所的研究人員正在與 NVIDIA 一起使用 BioNeMo 框架來開發新一代 DNA 語言模型。這些模型將被整合至 Terra 中。Terra 是一個由博德研究所、微軟和 Verily 共同開發的云平臺,生物醫學研究人員能夠通過該平臺,安全、大規模地共享、訪問和分析數據。這些 AI 模型還將被添加到 BioNeMo 服務集中。
OpenFold 聯盟計劃使用 BioNeMo 框架來推進其 AI 模型開發工作。這些模型可根據氨基酸序列來預測分子結構,并達到接近實驗的準確性。
Peptone 專注于構建固有無序蛋白質(缺乏穩定 3D 結構的蛋白質)的模型。該公司正與NVIDIA 一起使用同樣作為 BioNeMo 基礎的 NeMo 框架來開發 ESM 模型版本。該項目計劃在 NVIDIA 的 Cambridge-1 超級計算機上運行,將推動 Peptone 的新藥研發工作。
位于芝加哥的生物技術公司 Evozyne 將工程與深度學習技術相結合,致力于設計能夠解決長期治療和可持續發展難題的新型蛋白質。
Evozyne 聯合創始人兼計算主管 Andrew Ferguson 表示:“BioNeMo 框架使我們能夠在設計-構建-測試周期內,高效利用 LLM 的力量,基于數據進行蛋白質設計。這將直接影響我們新型功能性蛋白的設計,并將在人類健康和可持續發展方面得以應用。”
NVIDIA 初創加速計劃成員、生物技術初創企業 Peptone 的機器學習負責人 Istvan Redl 表示:“隨著大型語言模型在蛋白質領域的應用日益廣泛,高效訓練 LLM 并快速調整模型架構的能力變得非常重要。我們相信 BioNeMo 框架將滿足可擴展性和快速實驗這兩大工程方面的需求。”
審核編輯:湯梓紅
-
NVIDIA
+關注
關注
14文章
5021瀏覽量
103261 -
AI
+關注
關注
87文章
31133瀏覽量
269470 -
生物學
+關注
關注
0文章
26瀏覽量
12988
原文標題:GTC22 | NVIDIA 將大型語言模型擴展到生物學領域
文章出處:【微信號:NVIDIA_China,微信公眾號:NVIDIA英偉達】歡迎添加關注!文章轉載請注明出處。
發布評論請先 登錄
相關推薦
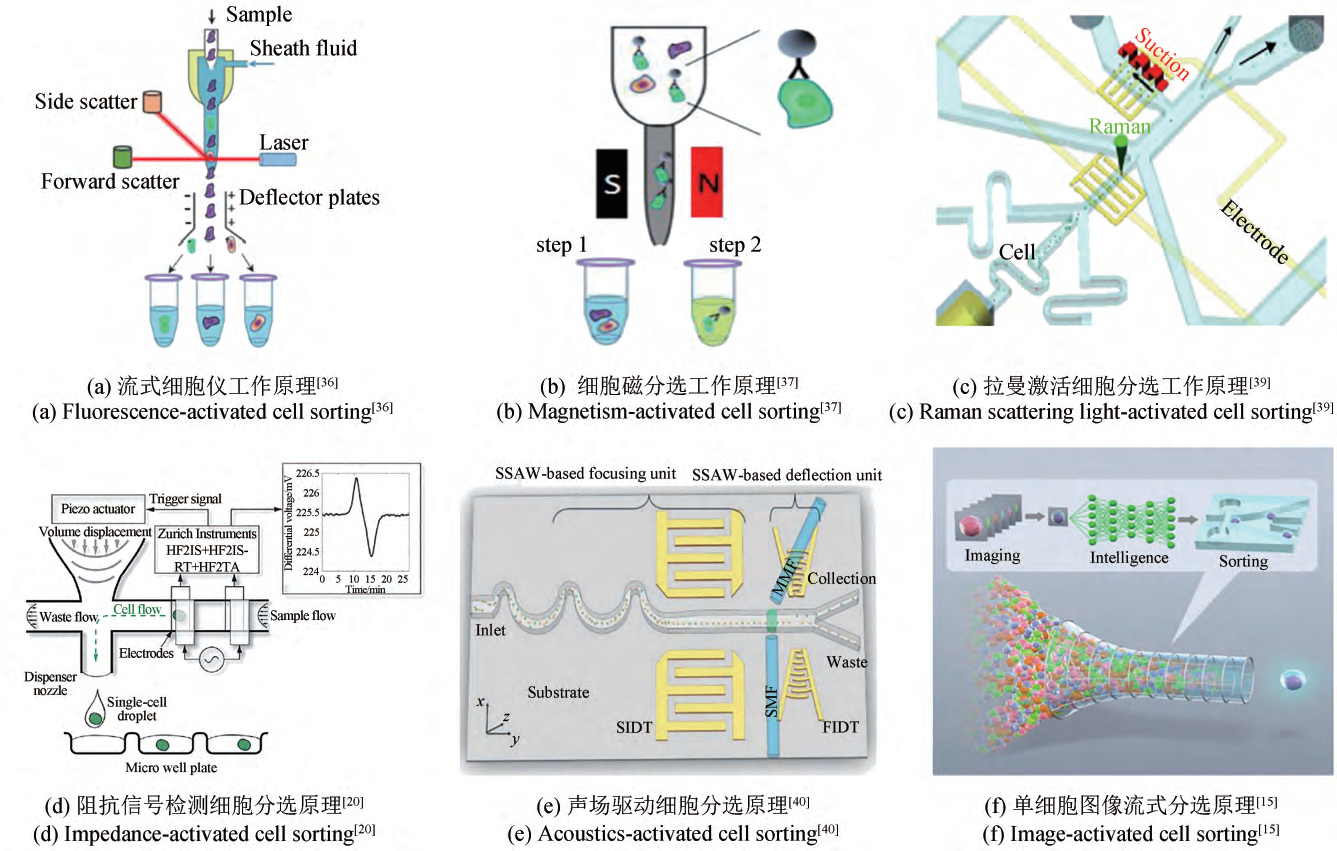
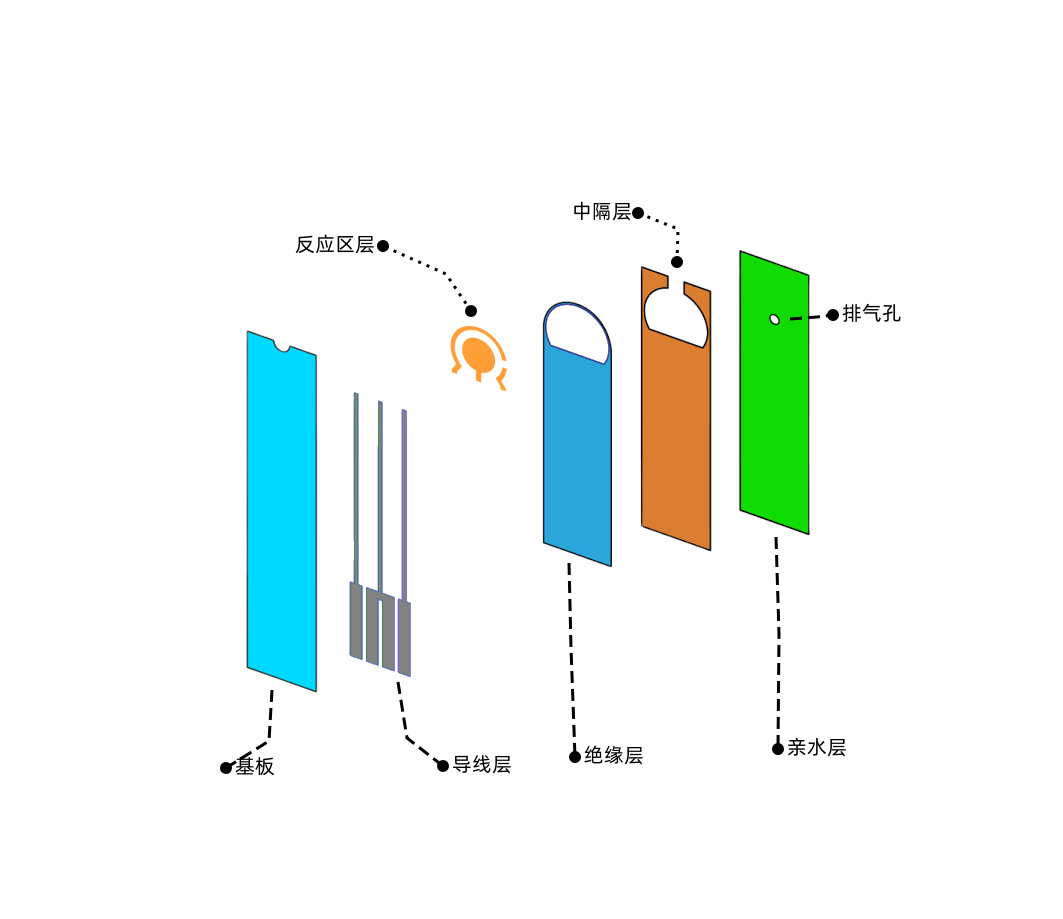
微流控技術的生物學應用
NVIDIA 推出 BioNeMo 開源框架,擴大全球生物制藥和科學行業的數字生物學研究規模
《AI for Science:人工智能驅動科學創新》第一章人工智能驅動的科學創新學習心得
ADI與與生物平臺創新公司Flagship Pioneering達成戰略合作
NVIDIA Omniverse 將為全新 OpenPBR 材質模型提供原生支持
借助NVIDIA DOCA 2.7增強AI 云數據中心和NVIDIA Spectrum-X
是德科技與NVIDIA攜手推進6G研究云平臺發展
Cadence與NVIDIA聯合推出利用加速計算和生成式AI重塑設計
基于NVIDIA開源CUDA-Q量子計算平臺發布
NVIDIA 推出云量子計算機模擬微服務





 NVIDIA BioNeMo框架將為生物學研究提供加速
NVIDIA BioNeMo框架將為生物學研究提供加速











評論